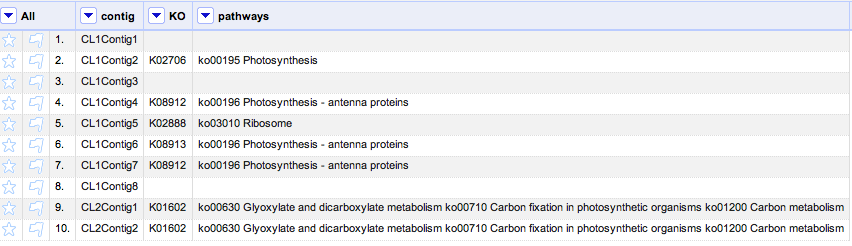
ちょうどNARに論文が公開されて、良い時期なのではないかと思い作成しました。
いつもはRAの学生さんがtogotvを作成されていますが、ヘビーユーザーがtogotvを作成すると面白いのではないかと考えたからです。
cyanobaseの特徴は何か?ということが伝わるように考えました。
1番の特徴はマニュアルキュレーションにより文献から抽出した情報がDBで公開されていることです。
文献から抽出した情報のうち、最も使われているのは
Gene symbol extracted from literature
だと思います。
これは他のDBでは公開されていない、cyanobase独自のものです。
TogoAnnotationを利用し、専門知識を持ったキュレーターが論文から手動で情報を抽出したものです。
時折誤った情報が紛れていますが、これは論文著者自身のミスが少なからず含まれています。
キュレーターが修正する方法もありますが、著者自身の記述を優先するという方針で入力しています。ご了承下さい。
遺伝子に対する参考文献情報も充実しております。
参考文献情報では、その遺伝子について言及しているセクション情報や同じ論文で記述されている他の遺伝子の情報を見ることが出来ます。
是非ご利用ください。