たとえば、
/cyanobase/Synechocystis/genes/slr1311 は遺伝子ID slr1311 のページを提供しています。
調べたい遺伝子IDを入力しなくても、直接そのページに移動する事が可能です。
これを利用して、遺伝子のリストを作成し、すぐにその遺伝子のページに移動できるようにGoogle Spread Sheetを使ってみます。
Google Driveの使い方はTogoTVを参照して下さい。
遺伝子IDのリストをGoogle Spread Sheetに入力します。
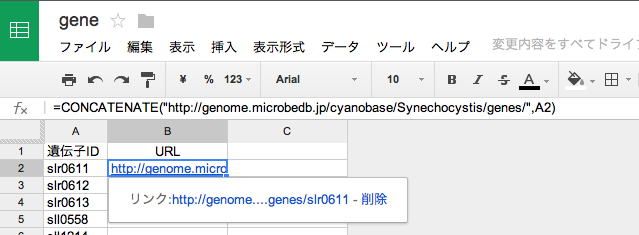
列Bにそれぞれの遺伝子IDに対応したURLを生成します。
関数は CONCATENATEを利用しています。 文字列を別の文字列に結合します。
詳しくは以下のページを参照して下さい。
URLを指定する"http://genome.microbedb.jp/cyanobase/Synechocystis/genes/"という文字列と、遺伝子IDが入力されているセルA2の文字列を結合します。
これで、リンク先のURL http://genome.microbedb.jp/cyanobase/Synechocystis/genes/slr0611 が生成され、すぐに遺伝子の情報のページを見ることができます。
この関数はGoogle Spread Sheedだけでなく、Excelでも使う事が出来ます。